データの分布の様子を見るには箱ひげ図を使用することが広く知られていますが、バイオリンプロットという作図方法でも可視化することができます。
バイオリンプロットは知名度は低いですが、箱ひげ図よりも多くの情報を表示できるので、作図する事例もそこそこあるようです。
バイオリンプロットはpythonやRだと専用のパッケージがあるのですが、SASにはそのようなものはないため、手動で作図する必要があります。
バイオリンプロットとは
バイオリンプロットはデータの分布をカーネル密度推定(kernel density estimation 以下KDE)を用いてノンパラメトリックでデータの分布を推定し、
分布関数を中心線をはさんで左右対称に表示する作図方法です。
多峰性(multimodal、つまり分布の山が複数ある)の分布や一様分布のデータを箱ひげ図で作図すると、その特徴が表現できませんが、バイオリンプロットは分布の特徴を正確に表現することができます。
GTLではバイオリンプロットを作図するステートメントはありませんので、一度KDEを実行して推定した密度を既存のグラフステートメントで作図する方法が考えられます。
バイオリンプロットの作図方法は公式ブログにも紹介されています。sgplotで作図は可能ですが、ここではこのブログ記事を参考にGTLで作図します。
SASでKDEを実行する。
SASでKDEを実行するにはproc kdeを使用します。
KDEを実行するにはバンド幅というパラメータをデータに応じて設定する必要がありますが、SASではデフォルトでは Sheather-Jones plug-in method という方法でバンド幅を自動決定してくれるので、こだわりがなければバンド幅の設定は不要かと思います。
ここではsahelp.heartから各死亡理由毎に血中コレステロール濃度の分布を推定します。
*データの抽出;
data cholesterol;
keep deathcause cholesterol;
set sashelp.heart;
if deathcause ^= '';
proc sort; by deathcause;
run;
* kde;
proc kde data = cholesterol;
by deathcause;
univar cholesterol / out = density ;
run;proc kdeで1変数分布を推定する場合はunivarステートメントを使用して推定したい変数を指定します。
2変数分布を推定したい場合はbivarステートメントを使用します。
byステートメントを使用しサブグループ毎(ここではdeathcause)を実行します。
Deathcause=cancerの実行結果は以下の通りです。
| 入力 | |
|---|---|
| Data Set | WORK.CHOLESTEROL |
| Number of Observations Used | 521 |
| Variable | Cholesterol |
| Bandwidth Method | Sheather-Jones Plug In |
| コントロール | |
|---|---|
| Cholesterol | |
| Grid Points | 401 |
| Lower Grid Limit | 60.25 |
| Upper Grid Limit | 417.75 |
| Bandwidth Multiplier | 1 |
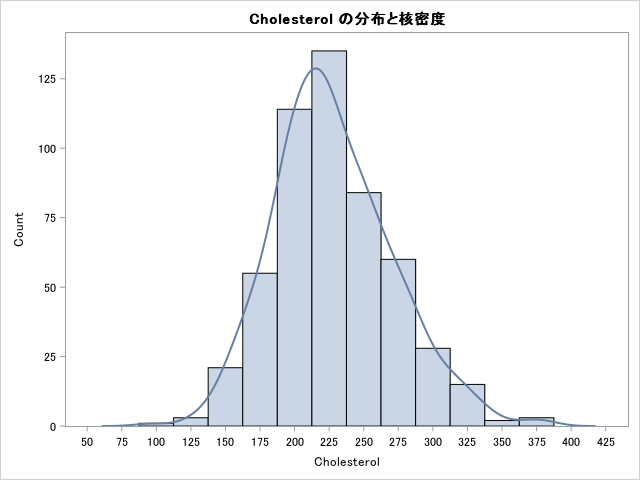
推定結果(死亡理由=cancer)
推定結果をヒストグラムと一緒に図示してくれるのは便利ですね。
出力データセットは以下の通りです
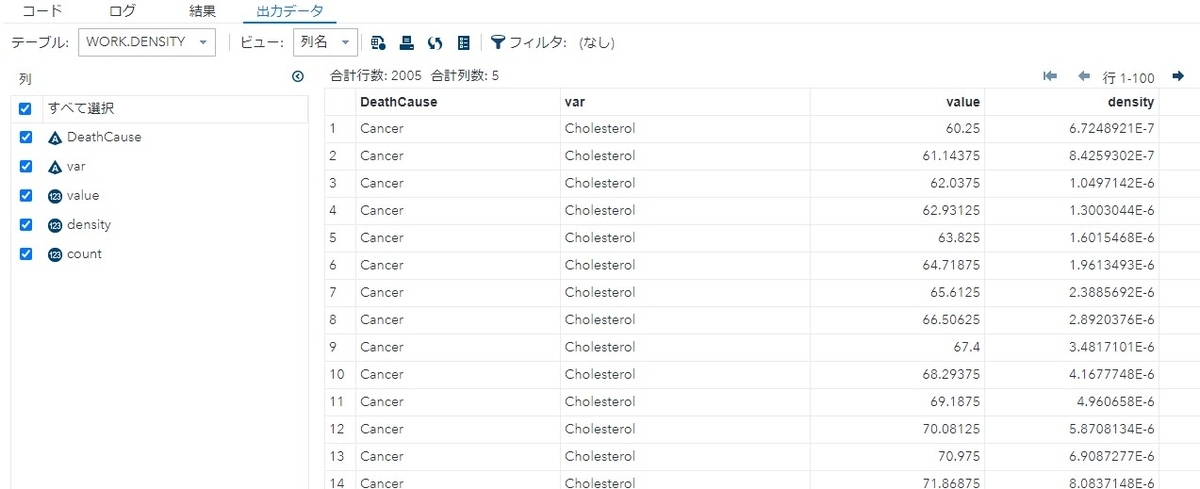
。
var=密度推定したい変数
value=変数の値(ここでは血中コレステロール濃度)
density=valueに対応する密度
count=読み込んだオブザベーションから集計された度数
バイオリンプロットはこのdensityを利用して作図します。
次回はGTLで作図する方法を紹介します。






