前回はアゴニスト濃度と生体の応答の関係性を非線形回帰分析にて推定しました。
今回は推定したパラメータを用いて濃度応答曲線(concentration-response curve あるいはdose-response curve)を作図します。
nlinプロシジャのplotオプションを使えば自動でこの作図をやってくれますが、ちょっとがたがたしてて滑らかな曲線ではないし、論文用の図表としてカスタマイズしたいケースもあるので、作図データセットを作成してGTLで作図します。
予測値と信頼区間の出力
nlinプロシジャのoutputステートメントで推定したパラメータから予測される応答とその信頼区間を出力できます。
前回のデータを用いて以下のコードを実行するとpredデータセットには入力データセットの変数に加えて、パラメータ推定値とその信頼区間と予測区間を「統計量=変数名」の形で出力します。
なおparmsオプションではパラメータ名以外の変数名も指定できますが、その場合はparametersステートメントにて指定した順番とparmsオプションで指定する順番は合わせましょう。
proc nlin data=raw ;
parameters / pdata=pdat;
model response=bottom+(emax-bottom) / (1+10**((logec50-conc)*slope));
output out=pred
parms=emax bottom logec50 slope
pred=estimate
uclm=uclm
lclm=lclm
ucl=ucl
lcl=lcl;
by type;
run;
- parms: 推定したパラメータ 複数ある場合はスペースで区切る
- pred: 予測値
- lclm: 下側信頼限界
- uclm: 上側信頼限界
- lcl: 下側予測限界
- ucl: 上側予測限界
信頼区間および予測区間はalphaオプションを指定しない限り95%となります。
predデータセットには濃度と予測値が格納されたので、これを使ってsgplotで作図すると・・・
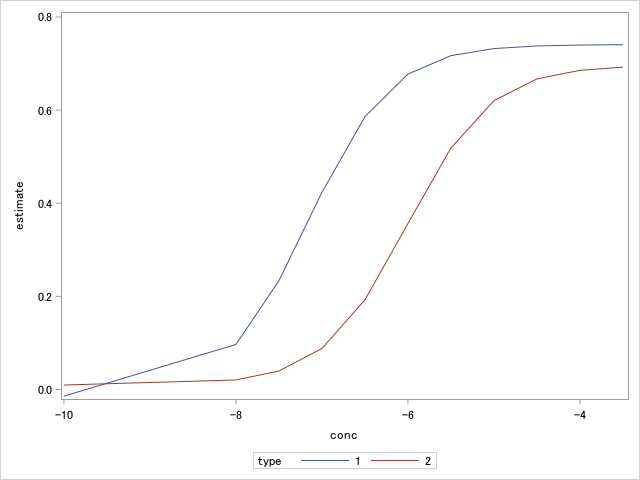
かなりカクカクしたグラフが出来上がります。あくまで入力した濃度に対する予測値しか出力されていないので、その間は直線で結ばれます。
本来は滑らかなシグモイド曲線なのですが・・・
そこでnlinプロシジャに入力するデータを少し改良します。
作図用データセットの作成
nlinプロシジャは入力した説明変数の値に対する予測値した出力しないので、入力データを増やせば曲線のポイントが増えて滑らかになります。
今回は対数濃度を-10から-3.5まで0.02刻みで格納します。測定していない濃度の応答値は当然欠損値となり、パラメータ推定の計算では無視されます。予測値を算出するときは各濃度に対応する予測値を出力してくれます。
濃度系列を格納したダミーデータを測定結果データセットにマージし、それを上記のコードで解析します。出力されたpredデータセットを作図すると以下のようになります。
data dmy;
do type=1 to 2;
do conc= -10 to -3.5 by 0.02;
output;
end;
end;
run;
proc sort data=raw; by type conc;
run;
/*作図データセットの作成*/
data raw2;
merge raw dmy;
by type conc;
run;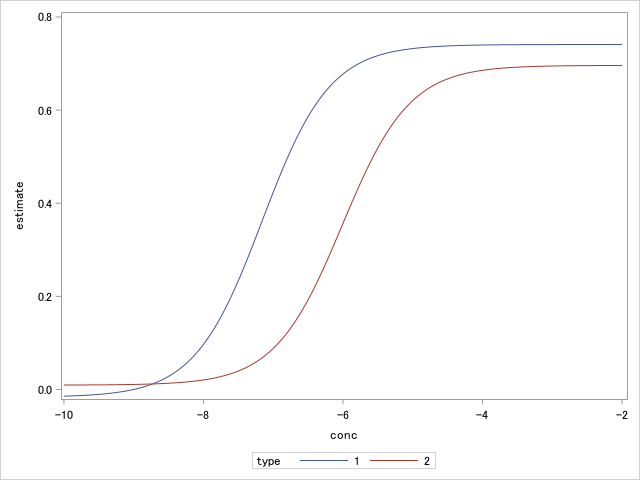
滑らかな曲線を作図できました。SASの作図は数式をそのまま作図する機能はないようなので、作図用のデータを生成する必要があります。滑らかな曲線を出力するためにはレコードをたくさん用意する必要があります。どれくらい必要なのかは作図してみて決めると良いです。まあデータが多少多くても実行速度にはさほど影響はないので、今回は設定した濃度の範囲を細かく分割しました。
発表用の作図
発表用の資料としてもう少し手を加えます。sgplotでもできますが、今回はGTLのGRIDDEDレイアウトを利用してグラフ内にパラメータの推定値を載せます。typeにもちゃんとフォーマットをあててわかりやすくしましょう。
グラフ内テーブルはOVERLAYレイアウト内にGRIDDEDレイアウトを設置して作成します。

パラメータ推定値は有効数字3桁で表示します。自作関数があるのでこれを使います。

これを作ってて気づいたのですが、ベースライン補正しているのでモデルのbottomは0で固定ですね。推定する必要はありませんでした。モデルを修正します。
model response=emax / (1+10**((logec50-conc)*slope));
またアゴニスト濃度がmol/Lのままだと桁数が多すぎなのでnmol/Lに変換することにします。
/*フォーマット作成*/
proc format;
value type 1="Treated"
2="Non treated";
run;
/*推定値をマクロ変数に格納*/
data pred;
length emax_1 ec50_1 emax_2 ec50_2 $20;
set pred;
by type;
if first.type then select (type);
when (1) do;
call symputx ("emax_1" ,sig(emax,3));
call symputx ("ec50_1" ,sig(10**logec50,3));
end;
when (2) do;
call symputx ("emax_2" ,sig(emax,3));
call symputx ("ec50_2" ,sig(10**logec50,3));
end;
end;
run;
proc template ;
define statgraph curve;
dynamic _emax_1 _emax_2 _ec50_1 _ec50_2;
begingraph;
entrytitle "被験物質処理による細胞の応答変化";
layout overlay /
xaxisopts=(label="Log Agonist concentration (nmol/L)")
yaxisopts=(label="Response");
seriesplot x=conc y=estimate /
group=type
name="series";
scatterplot x=conc y=response /
group=type
name="scatter"
markerattrs=(symbol=circlefilled);
layout gridded /
autoalign=(TOPLEFT TOPRIGHT) columns=3 border=true;
entry "Parameter" / textattrs=(weight=bold) pad=5 ;
entry "Treated"/ textattrs=(weight=bold) pad=5 ;
entry "No treated"/textattrs=(weight=bold) pad=5;
entry halign=left "E"{sub "max"};
entry _emax_1;
entry _emax_2;
entry halign=left "EC"{sub "50"};
entry _ec50_1;
entry _ec50_2;
endlayout;
mergedlegend "series" "scatter";
endlayout;
endgraph;
end;
run;
/*マクロ変数をdynamic variableに代入*/
proc sgrender data=pred template=curve;
format type type.;
dynamic _emax_1=&emax_1
_emax_2=&emax_2
_ec50_1=&ec50_1
_ec50_2=&ec50_2;
run;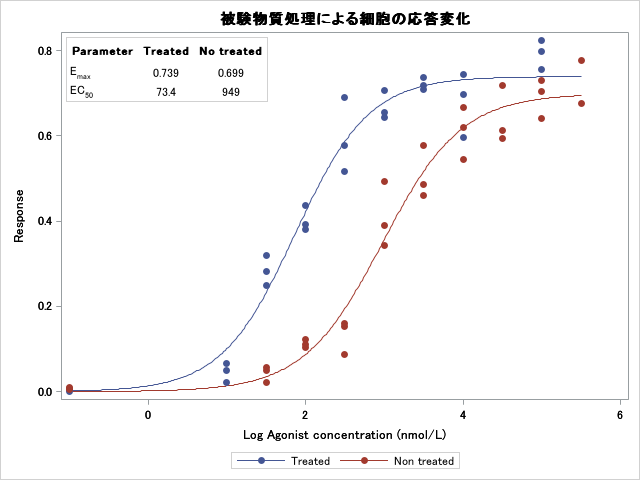
被験物質を処理することによってEC50が10分の1以下になっていることがわかります。
これなら発表用としても十分でしょう。


