最近duckdbを使うことが増えてきたので備忘録としておいておきます。
ビックデータ分析においてRやpythonだとどうしても処理速度に難があるため、あらかじめRAWデータを要約してから用いると効率的です。最近はDuckDBやClickHouse といったオープンソースで使えるOLAP(Online analytical processing)が注目を集めており、手軽に分析用データベース環境を構築できるようになりました。今後医療データの大容量化に伴い医療データ分析もOLAPへ移行する可能性は大いにあると思います。
今回はDuckDBをRstudioにセットアップする方法を紹介します。
導入方法
他のパッケージと同様にduckdbのパッケージをインストールするだけです。他に依存するソフトウェアが不要なのもメリットですね。
#パッケージのインストール
install.packages("duckdb")
#必要なパッケージの読み込み
library(duckdb)
library(tidyverse)
library(arrow)
library(DBI)
library(connections)使用前にDBIパッケージを用いてduckdbデータベースの作成と接続を実施します。この時dbdirパラメータにデータベースを作成するディレクトリを指定するのですが、メモリ上にも作成することができます。
ただし個人的にはディレクトリ上に作成することをお勧めします。
一般的なPCのメモリは多くて16GBくらいです。レコード数数千万から1億くらいのデータの場合、中間テーブルを作成するとメモリが足りなくなる可能性が高いです。それにディスク上にデータベースを作成してもRよりは断然高速に処理できます。
大容量メモリを搭載したマシンで実施するのであればメモリ上に作成すると良いと思います。
#メモリ上にデータベースを作成する。
con <- dbConnect(duckdb(), dbdir = ":memory:")
#ディレクトリ上にデータベースを作成する
con <- dbConnect(duckdb(), dbdir = "my-db.duckdb")
データの入出力
データの入出力およびクエリの実行はDBIの関数を使用します。
今回はgoogleのCOVID-19オープンデータの疫学データ(epidenmiology.csv)をduckdb上に展開してみます。
オープンデータはcsvまたはjson形式で提供されています。duckdbはどちらの形式でもテーブルの操作と同じように直接読み書きすることができます。csvファイルを直接テーブルとしてデータベースに登録する場合は以下のクエリを実行します。
# dbExecute:データベースにクエリを送信する
# dbExecute(データベース接続, "クエリ")
dbExecute(con,"create or replace table raw as select * from 'epidemiology.csv'")
なお分析時はクエリの修正と実行を繰り返していろいろ検討することになると思いますが、create tableステートメントだとテーブルの上書きは許容されていないため、再実行時にすでにテーブルが存在するとエラーになります。create or replace tableステートメントにすると既存のテーブルが存在する場合は上書きできるようになります。分析目的でSQLを実行する場合はこっちのほうが便利。
# 疫学データの日付を取り出す
dbExecute(con,"
create or replace table raw as
select distinct date
from raw;
")
テーブルをR環境に出力する場合は、dbReadTable関数を用います。dbReadTableを用いるとテーブルはdata.frame形式で出力されます。
Rのデータ前処理の必須パッケージであるtidyverseはtibble形式が標準であるため、念のためtibble形式に変換しています。
# dbReadTable:データベースのテーブルをdata.frame形式でR環境に出力する
# dbReadTable(データベース接続, "テーブル名")
raw<-tibble(dbReadTable(con,"raw"))
str(raw)
tibble [12,525,825 × 10] (S3: tbl_df/tbl/data.frame)
$ date : Date[1:12525825], format: "2020-01-01" "2020-01-02" "2020-01-03" "2020-01-04" ...
$ location_key : chr [1:12525825] "AD" "AD" "AD" "AD" ...
$ new_confirmed : num [1:12525825] 0 0 0 0 0 0 0 0 0 0 ...
$ new_deceased : num [1:12525825] 0 0 0 0 0 0 0 0 0 0 ...
$ new_recovered : num [1:12525825] NA NA NA NA NA NA NA NA NA NA ...
$ new_tested : num [1:12525825] NA NA NA NA NA NA NA NA NA NA ...
$ cumulative_confirmed: num [1:12525825] 0 0 0 0 0 0 0 0 0 0 ...
$ cumulative_deceased : num [1:12525825] 0 0 0 0 0 0 0 0 0 0 ...
$ cumulative_recovered: num [1:12525825] NA NA NA NA NA NA NA NA NA NA ...
$ cumulative_tested : num [1:12525825] NA NA NA NA NA NA NA NA NA NA ...
ローカルファイルとしてテーブルを出力する場合はsqlのcopyステートメントを使います。出力形式はcsv、json、excelなどが利用できますが、今回は汎用性の高いparquet形式で出力してみます。
dbExecute(con,"copy raw to 'output.parquet' (format parquet);")
個人的にはarrowパッケージを使ったほうが圧縮率がいいような気がしているのでparquet形式の変換はarrowパッケージのwrite_parquet関数を用いています。私はduckdbでデータ前処理、要約を実施した後に、最後にR環境に出力しSQLでできない処理を実施していますので、parquet形式の出力はarrowパッケージのみつかっています。
#tibbleをparquet形式で書き出す
arrow::write_parquet(テーブル形式のデータ, sink="ファイル名")
データベースの内容を確認する
Rstudioはもともと外部データベースへの接続をサポートしており、GUIでデータベースの情報を確認できるようになっています。connection_view関数で内容を確認したいデータベース接続を指定すると、Rstudioの右上に表示されているConnectionペインに指定したデータベースの情報を確認することができます。
先ほど作成したテーブルrawがあることが確認できます。
connections::connection_view(con)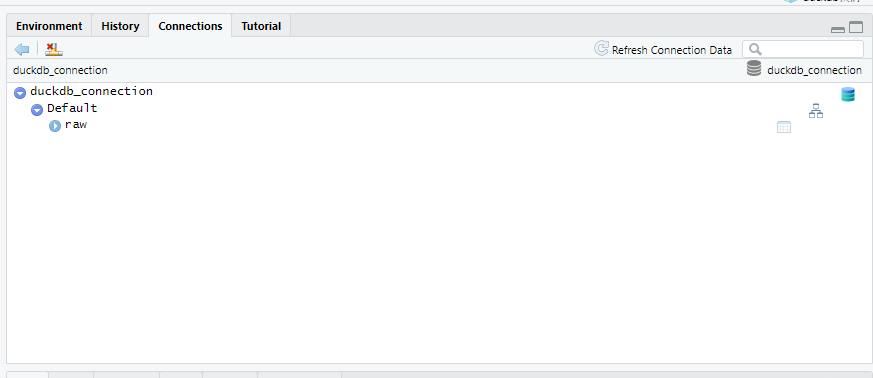
テーブルのカラム情報も確認でき、さらに右側のテーブルのアイコンをクリックすれば直接テーブルの中身も確認することができます。これは便利。
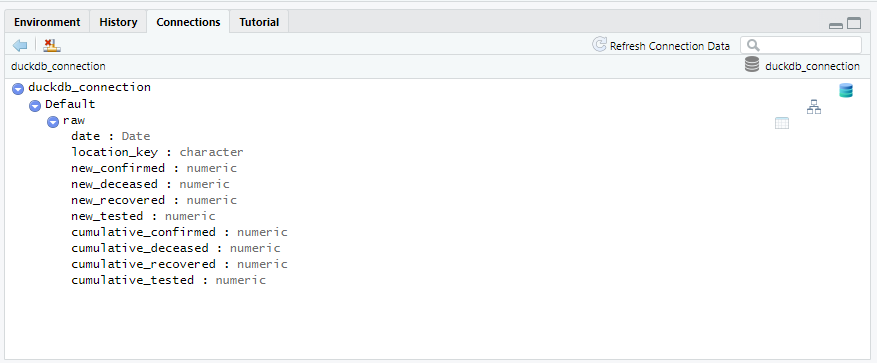
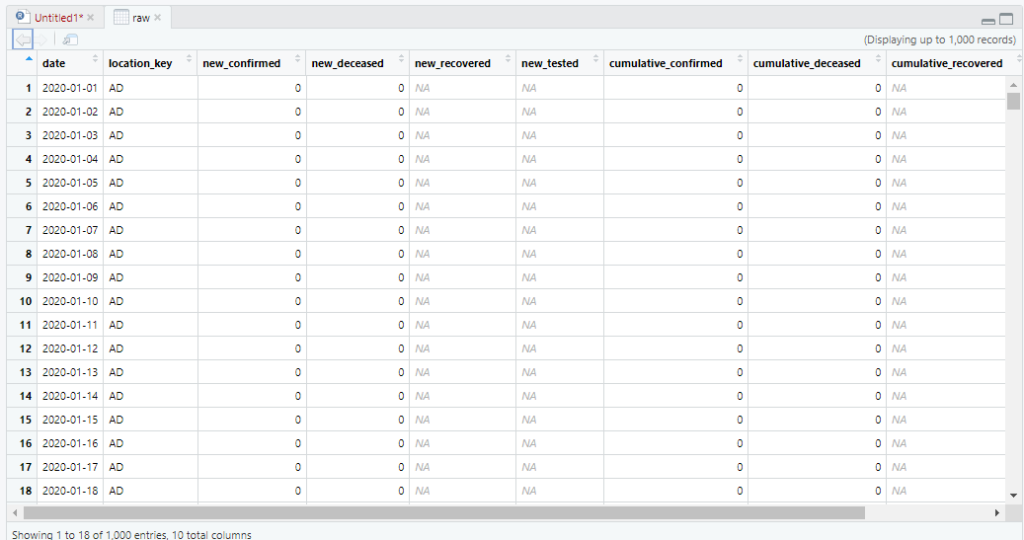 データベースのテーブルの中身を直接確認する
データベースのテーブルの中身を直接確認する



