ggplot2を使えばヒストグラムは自動で作成できますが、集計済みのデータが必要になるケースがあります。特にダブルプログラミングによる検証をするつもりなら、グラフの元となる集計済みデータが欲しくなりますよね。
今回はヒストグラム作成に必要なデータ処理であるビニングとその頻度集計を実施してみます。
グラフを作りだけなら簡単だけど。。
ggplot2のgeom_histogram()関数を使えばビニングを実施しなくても自動で集計してグラフにしてくれます。
library(tidyverse)
iris %>%
ggplot(aes(x=Petal.Width,fill=Species)) +
geom_histogram()
ヒストグラムの結果をテーブルとして表示したい場合や、検証のために集計結果を確認したい場合はヒストグラムの画像だけでなく作図のもとになったデータも欲しくなります。
手作業で集計するのも良いですが、ggplot2では集計済みデータの取得できるようになっていますので、それを利用するのが一番簡単です。
作図用データの取得
グラフ作成時に作成したデータはggplot_build()関数で取得できます。先ほどのヒストグラム作成時に生成したggplotオブジェクトをggplot_build()関数に渡すと以下のようなリストを返してくれます。
gobj <-iris %>%
ggplot(aes(x=Petal.Width,fill=Species)) +
geom_histogram()
gdat <- ggplot_build(gobj)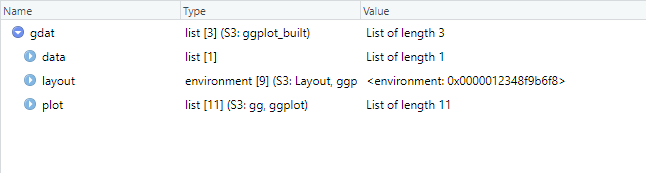 ggplotオブジェクトの中身
ggplotオブジェクトの中身作図の座標データだけでなく色コードや軸の範囲も出力されるようです。
gdatはリストになってますからここから必要なデータを取り出しても良いですが、今回の場合は作図の座標データのみが欲しいので、layer_data関数を使ったほうが楽です。この関数は作図の座標データを直接返します。
coord <- layer_data(gobj)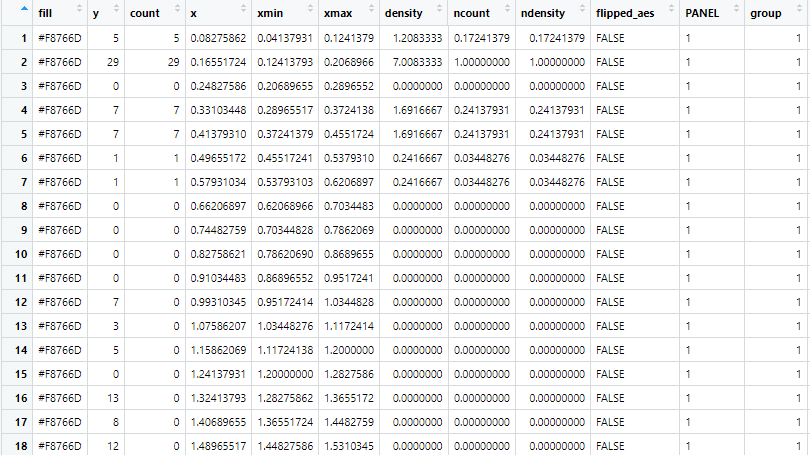 データフレームcoordの中身
データフレームcoordの中身xがビンの中心(midpoint)、xminとxmaxはビンの境界、countは対応するビンの頻度となります。ここからグラフ作成の元となった頻度集計結果がわかります。ただしこのデータだと頻度はわかりますが、各群の割合は算出してくれないようです。(densityは割合ではないです)
複数のヒストグラムを比較する場合は頻度ではなく割合で見たほうがわかりやすいと思いますので、マニュアルで割合を算出してみましょう。
ビニングと集計
ビニングの関数はいくつかありますが、今回はggplotのcut_width()関数を使います。
cut_width()関数はビン幅を指定してビニングしてくれます。Rのcut関数もビニングを実行する関数ですが、ggplotの関数のほうが便利。ビンの数を指定したい場合はcut_interval()関数を使うと良いでしょう。
引数widthでビン幅、引数centerでビン中点を設定します。centerの意味が分かりにくいですが、実際の設定例を見ればイメージできると思います。
例えば今回のデータでwidth=0.2と設定すると、ビンは
[0.1, 0.3], (0.3, 0.5] …
になります。最初のビンの左側境界はデータの最小値に指定されます。width=0.2, center=0.1と設定すると
[0, 0.2], (0.2, 0.4] …
になります。なお丸かっこは開区間(境界を含まない)、角かっこは閉区間(境界を含む)となります。
bin.wk1 <- iris %>%
dplyr::mutate(
bin=cut_width(Petal.Width,width=0.2, center=0.1)
)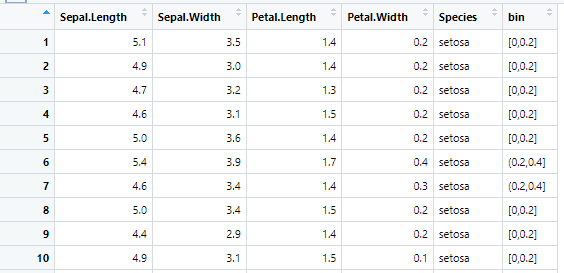 ビニング結果
ビニング結果
後はビニング結果に基づいて種別に集計するだけです、dplyrだけでOK。
bin.wk1 <- iris %>%
#ビニング
dplyr::mutate(
bin=cut_width(Petal.Width,width=0.2, center=0.1)
) %>%
#頻度集計
dplyr::group_by(Species, bin) %>%
dplyr::summarise(
count=n(), .groups="keep"
) %>%
#分母と割合の導出
dplyr::group_by(Species) %>%
dplyr::mutate(
countT=sum(count),
pct=count / countT * 100
) %>%
dplyr::ungroup()
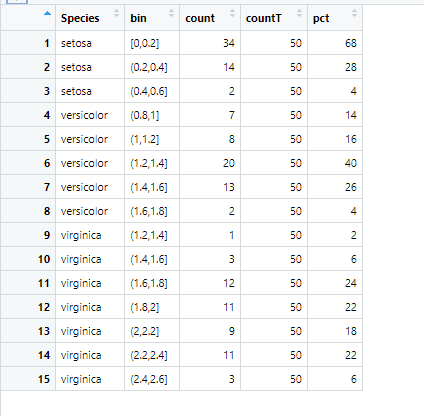 ビニングの集計結果
ビニングの集計結果
summariseはグループ変数に基づいてレコードが集約されるのに対し、mutateは集約されないという違いがあります。このデータフレームからテーブルも作成できるし、グラフも作成できるはずです。
次回はこのデータからヒストグラムを作ります。