
今年最初の記事はRでいきます。
去年から本格的にR, pythonを使い始めて、簡単な操作ならSASでなくてもできるようになってきました。
今回は統計の初歩かつ基礎となる頻度集計を実施してみます。
有害事象の発生状況を集計する
有害事象の発生状況の集計は治験や製販後調査において必ず実施する項目です。
今回は有害事象データを重篤度別に集計してみます。
有害事象データはMedDRAでコーディングされているものとします。
有害事象はSOC(System Organ Class, 器官別大分類)とPT( Preferred Term, 基本語)毎に発現した症例数を集計します。
ただし同じ事象であっても異なる重篤度の事象が収集された場合、それぞれ1例として集計します。事象はAESOCIO昇順およびAEPTCD昇順で表示します。
なおSOCとPTは階層構造になっており、SOCに複数のPTが属する形になっています。
有害事象データの構造は以下の通り
| 列名 | 説明 |
|---|---|
| USUBJID | 被験者識別コード |
| AESOCIO | SOC国際合意順 |
| AESOC | SOC日本語 |
| AEPTCD | PTコード |
| AEPT | PT日本語 |
| AESER | 重篤度(1=非重篤, 2=重篤) |
前処理
まず変数型を指定した上でExcel形式の有害事象データをインポートします。
変数の型は自動設定でも良いのですが、意図しない型推定を防ぎたいので私はすべての変数を文字列型として指定した上で取り込んでいます。
後で型変換しないといけませんが、この方法が一番安全かなと考えています。
症例数ですのでSOC, PT, 重篤度毎にUSUBJIDの重複を削除します。
重篤度別に加えて全体の集計も実施したいので、AESERをすべて3に置換したデータも作成して縦積みします。
SASはデータステップで一度変数を作成すると途中で変数の型を変更はできないのですが、Rの場合はカラム作成後に型変換してもOKなんですね。
用意したテストデータの事象数が多すぎるので今回は一部のSOCのみを集計することにしました。
# 事象と重篤度毎にUSUBJIDの重複を削除 adae <- read_excel("ADAE.xlsx", coltypes="text") %>% filter(AESOCIO %in% c(1:4)) %>% mutate( AESOCIO=as.numeric(AESOCIO), AEPTCD=as.numeric(AEPTCD), AESER=as.numeric(AESER)) %>% distinct(AESOCIO, AEPTCD, AESER, USUBJID, .keep_all=TRUE) # AESER=3 は全体の集計、USUBJIDの重複を削除 total <- adae %>% mutate( AESER=3) %>% distinct(AESOCIO, AEPTCD, USUBJID, .keep_all=TRUE) #縦積みにする freq1 < -bind_rows(adae, total) # SOCの集計用 USUBJIDの重複を削除 ソートの都合上、AEPTCDを0に置換する soc <- freq1 %>% mutate( AEPTCD=0, AEPT="") %>% distinct(AESOCIO, AESER, USUBJID, .keep_all=TRUE) #集計用データ freq_data <- bind_rows(freq1, soc) %>% arrange(AESOCIO, AEPTCD, AESER)
SASとtidyverseのコードの対応はこんな感じです。pandasよりもSASに近い形で書けるので
SASユーザーなら何回か触ればすぐ使いこなせそうです。
| SAS | tidyverse |
|---|---|
| 変数作成 | mutate |
| proc sort | arrange |
| proc sort; nodupkey ; by キー変数 | distinct(キー変数, .keep_all=TRUE) |
| データセットの縦結合(setステートメント) | bind_rows(データフレーム名) |
頻度集計
パッケージを使う前にtidyverseのみで集計を実施してみます。
group_byと summariseを併用するだけです。レコード数のカウントだけでなく各種要約統計量も算出できるので、
SASプロシジャのproc meansに相当します。
result < - freq_data %>% group_by(AESOCIO, AESOC, AEPTCD, AEPT, AESER) %>% summarise(count=n()) %>% ungroup()
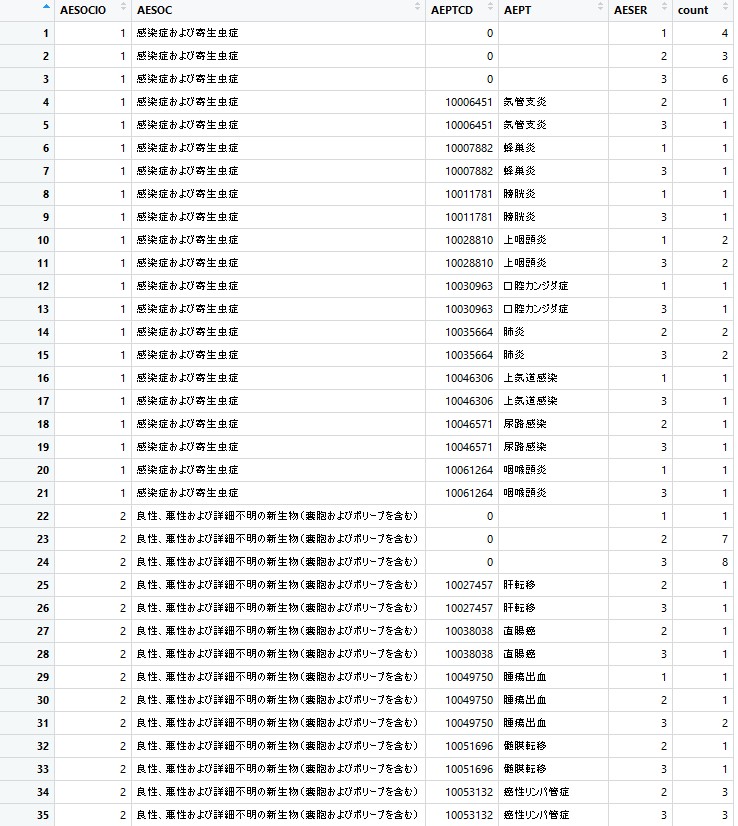
集計結果
集計結果を表にするのであれば、さらに加工する必要があります。
まずAESERに基づいて集計結果を横方向に並べます。SASだとproc transposeで実施している処理ですね。
この処理はtidyrのpivot_widerを使用すればOKです。転置後の列名はわかりやすい名前に変更します。
最後にNA(欠損)になっているセルを0で置換し、SOCとPTの表示を少し加工すれば完成です。
result_table <- result %>% #集計結果を横方向に展開 pivot_wider(names_from=AESER,values_from=count, names_prefix="COL") %>% # 欠損値を0で置換 replace_na(list(COL1=0, COL2=0, COL3=0)) %>% # PT集計行はSOC名を非表示にする mutate( AESOC=if_else( AEPTCD==0, AESOC,""), AEPT=if_else( AEPTCD==0, "",AEPT)) %>% 必要な列のみ選択 select(AESOC, AEPT, starts_with("COL")) %>% 列名変更 rename("非重篤"=COL1, "重篤"=COL2, "全体"=COL3)

集計結果
SASデータステップのif-elseステートメントは if_else関数または case_when関数で同様の処理が実現できます。
ここもSASに近い形で書けるのは良いですね。
このデータフレームをHTML形式にすれば成果物としてはOKでしょう。実際の成果物作成は次回実施します。
パッケージを使うべきか?
Rは便利なパッケージがたくさんありますが、当然頻度集計用パッケージもあります。tabylあたりが使えそうかなと思いましたが、今回のような層別の集計に対応していないみたいです。
つまり行あるいは列の変数を複数指定できません。これだったらtidyverseのみで集計したほうがよさそうです。集計によっては少々工夫が必要になりそうではありますが、自分で処理の流れを設計したほうが
後で検証しやすいと思うので、今後はtidyverseのみで集計していこうと思います。
あとRはSASと異なりパッケージの信頼性をどう担保するのかが課題になりそうです。
tidyverseはユーザー多いし開発規模も大きいですからまあ信用して問題ないと思いますが、それ以外のパッケージはよく検討したほうがよさそうです。
この点はSASのほうが優れていますね。





