生存時間分析は頻出の分析手法で、SASではLIFETESTプロシジャで実施します。
生存時間分析はカプランマイヤー法による解析が多いと思いますが、カプランマイヤー法により求めた生存率をプロットしたものがカプランマイヤー曲線(カプランマイヤープロット)です。
今回はそのカプランマイヤープロットのカスタマイズ方法を紹介します。
plotsオプション
LIFETESTプロシジャで生存率をデータセットとして出力し、それをsgplotで作図することもできますが、LIFETESTプロシジャのplotsオプションである程度カスタマイズしたグラフを出力することができます。
plots=survivalを指定すると自動的にカプランマイヤープロットが出力します。(LIFETESTプロシジャでは推定方法を指定しない場合デフォルトでカプランマイヤー法を使用します。)
まずはSASHELPファイルを使って実際に出力してみます。
proc format;
invalue $bmtifmt 'ALL' = 1
'AML-Low Risk' = 2
'AML-High Risk' = 3;
value bmtfmt 1 = 'ALL'
2 = 'AML-Low Risk'
3 = 'AML-High Risk';
run;
data Bmt2;
set sashelp.BMT(rename=(Group=G));
Group = input(input(G, $bmtifmt.), 1.);
label Group = 'Disease Group';
format Group bmtfmt.;
run;
proc LIFETEST data=Bmt2 cs="circle" plots=survival outsurv=survival;
time T*Status(0);
strata Group ;
run;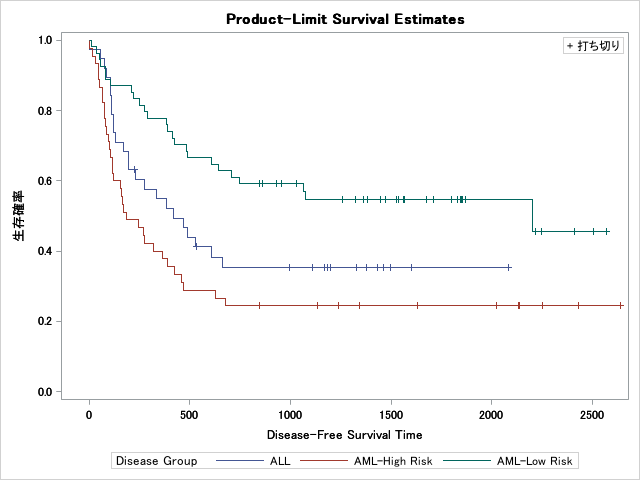
特に何も指定しない場合は生存率と打ち切り表示します。
打ち切りを非表示にする
打ち切りのマーカーを非表示にするにはplotsオプションにnocensorを追加します。
proc LIFETEST data=Bmt2 plots=survival(nocensor);
time T*Status(0);
strata Group / test=logrank;
run;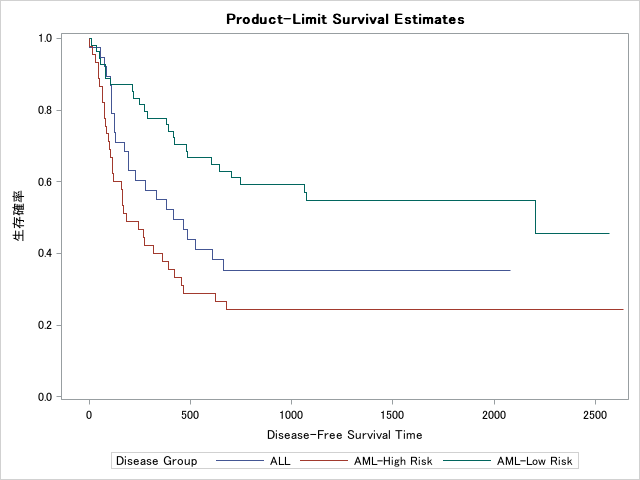
信頼区間を表示する
plotsオプションにclを追加すると信頼区間を表示します。デフォルトでは95% ですがalphaオプションで変更できます。例えば90%信頼区間を表示したい場合はalphaには0.1 (1-0.9)を指定します。
proc LIFETEST data=Bmt2 plots=survival(cl) alpha=0.1;
time T*Status(0);
strata Group / test=logrank;
run;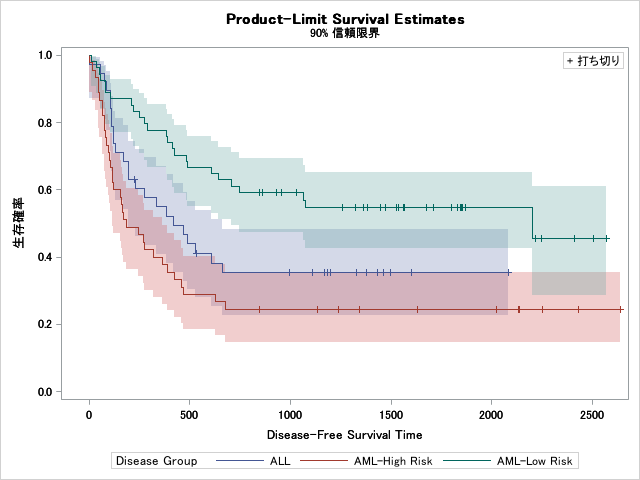
number at riskを表示する
plotsオプションにatriskを追加するとnumber at riskの表をプロットに挿入します。層ラベルはデフォルトでは12文字以上だと表示されません。その場合はmaxlenオプションでラベル文字数以上に設定するとラベルは正常に表示されます。
proc LIFETEST data=Bmt2 plots=survival(atrisk maxlen=25) ;
time T*Status(0);
strata Group / test=logrank;
run;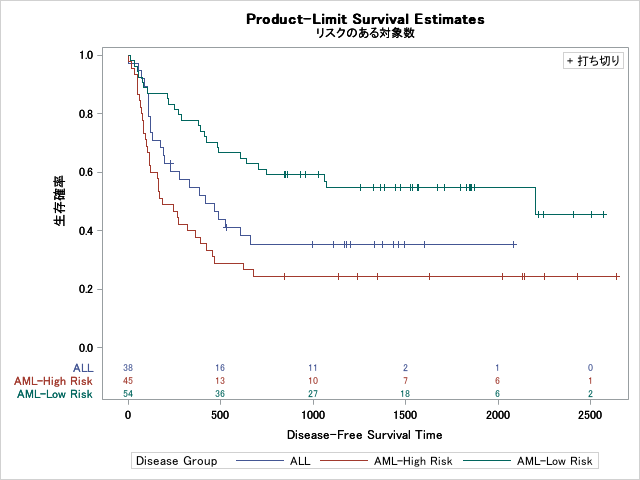
なおoutsideを追加するとnumber at riskの表は軸の下に配置されます。
proc LIFETEST data=Bmt2 plots=survival(atrisk maxlen=25 outside) ;
time T*Status(0);
strata Group / test=logrank;
run;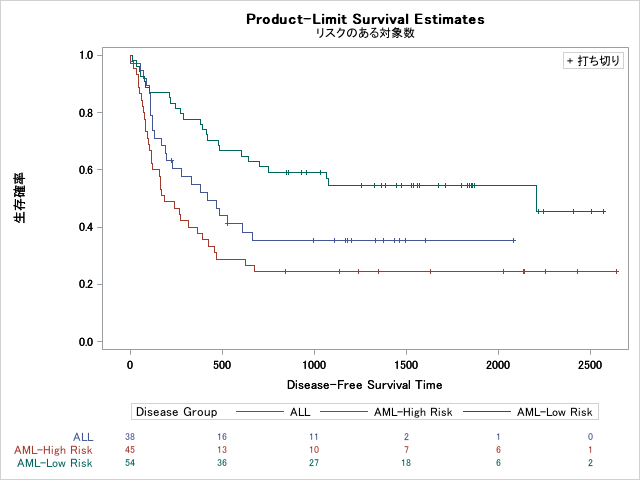
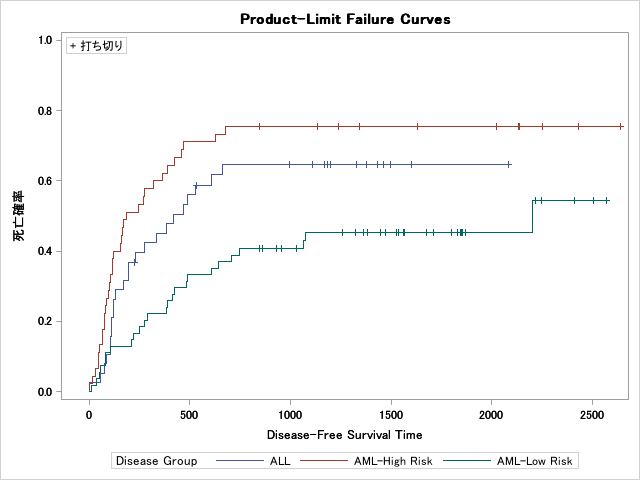
死亡率曲線(failure plot)を作図する
死亡率曲線(Failure plot)は単純に1から生存率を引いた値をプロットしたものです。イベント発生に着目している場合は作図することもあると思います。
生存曲線を加工しなくてもproc lifetestのオプションだけで出力することができます。
plotsオプションにfailureを指定するだけで、死亡率の変数を作成してくれます。
ods output failureplot=failure; proc LIFETEST data=Bmt2 plots=survival(failure);
time T*Status(0);
strata Group / test=logrank;
run;作図用データセットが欲しい場合はods output からfailure plotを取得すれば良いでしょう。
死亡率は変数「1_SURVIVAL__」, 打ち切りマーカーの位置は変数「1_CENSORED__」を利用することで
別の作図プロシジャで作図できます。
ログランク検定のp値を表示する
strataステートメントでクラス変数を指定した時、plotsオプションにtestを追加すると同質性の検定結果を凡例に表示できます。複数の検定を実施した場合は優先順位の高い結果のみを表示するようですが、基本的にログランク検定の結果が表示されると考えてよいでしょう。
proc LIFETEST data=Bmt2 plots=survival(test) ;
time T*Status(0);
strata Group / test=logrank;
run;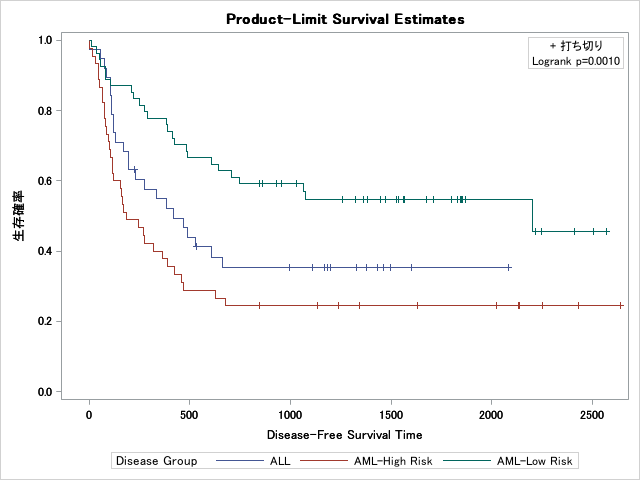
層ごとにプロットを分けて出力する
plotsオプションにstrata=設定値を指定すると層毎に分けてプロットを出力できます。設定値はoverlay panel unpackのいずれかです。panelはプロットエリアを分割してそれぞれの区画に生存率を表示します。unpackは層の数だけ上記の同様のグラフが出力されます。overlayは一つのプロットエリアにすべての層を重ねて表示します。
proc LIFETEST data=Bmt2 plots=survival(strata=panel) ;
time T*Status(0);
strata Group / test=logrank;
run;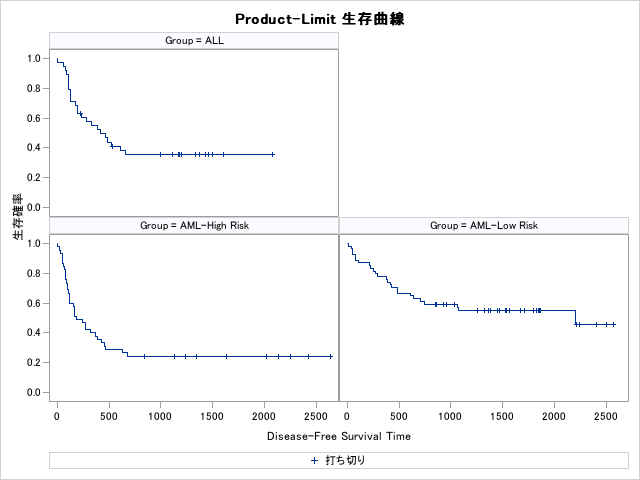
成果物としては物足りない
LIFETESTプロシジャだけである程度はカプランマイヤープロットを作成できますが、注釈や参照線の指定、プロット内テーブルに任意の統計量の表示等ができないので成果物としての提出はこれでは難しいです。
プロシジャの設定だけで要件を満たすことができればラッキーで、ほとんどの場合はGplotやsgplotを用いた作図は必要になってくるでしょう。



