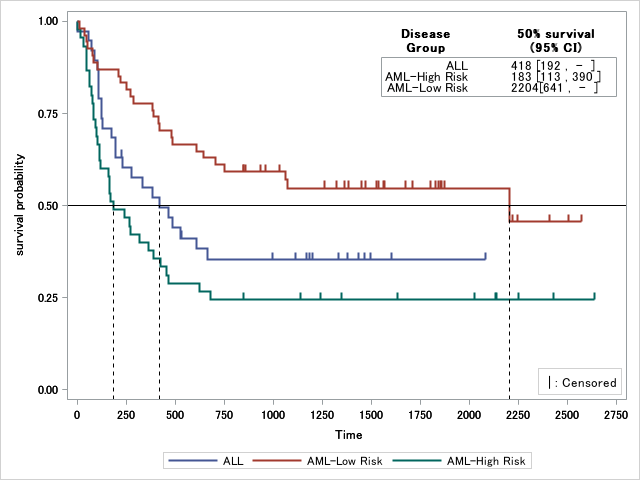
引き続きカプランマイヤープロットの作図について。今回はログランク検定や50%生存時間などの統計量を挿入し、参照線を挿入してデータをわかりやすく表示します。
生存時間中央値を表示する
生存率が50%になるのに要する時間(生存時間中央値: survival median time)を推定し、その推定値をプロットエリア内テーブルに表示します。
生存時間中央値はodsでQuartilesというデータセットを参照すると取得できます。なおalphaqtオプションで信頼区間の有意水準を変更できます。0.1に指定すると90%信頼区間が出力されます。
推定値と信頼区間を任意の文字列に成形し、軸変数用のダミー変数NOを作成した後、作図データセットであるsurvivalplotにマージします。
プロット内にテーブルを挿入するにはLAYOUT GRIDDEDを利用することが知られていますが、LAYOUT OVERLAYレイアウトを入れ子にしてaxistableを挿入する方法のほうが楽だと思いました。
GRIDDEDレイアウトだとentryステートメントをデータの個数だけ実行しないといけたいため、データが多いとその分コードが多くなります。
axisitableは必ずinnnermarginレイアウト内で使用するようにしましょう。
LAYOUT OVERLAYはブロックのサイズをwidth およびheightオプションで、配置位置をvalignとhalignで調整できます。あとはpadオプションで余白を調整すればOKです。
proc format;
invalue $bmtifmt 'ALL' = 1
'AML-Low Risk' = 2
'AML-High Risk' = 3;
value bmtfmt 1 = 'ALL'
2 = 'AML-Low Risk'
3 = 'AML-High Risk';
run;
data Bmt2;
set sashelp.BMT(rename=(Group=G));
Group = input(input(G, $bmtifmt.), 1.);
label Group = 'Disease Group';
format Group bmtfmt.;
run;
ods output survivalplot=plotdata
Quartiles=q;
HomTests=test;
proc LIFETEST data=Bmt2 plots=survival(atrisk(maxlen=24)=0 to 2750 by 250 outside) ;
time T*Status(0); strata Group ;
run;
*中央値の推定結果を加工する;
data q2;
set q(where=(percent=50));
length stat $50 est lower upper $4;
retain NO 0;
pct=percent/100;
if estimate^=. then est=strip(put(estimate,4.0));
else est=" - ";
if lowerlimit^=. then lower=strip(put(lowerlimit,4.0));
else lower=" - ";
if upperlimit^=. then upper=strip(put(upperlimit, 4.0));
else upper=" - ";
stat=cat(est,"[", lower, ", ", upper, "]");
NO+1;
keep NO group stat estimate pct;
run;
*データをマージする;
data plotdata2;
merge plotdata q2;
run;
proc template;
define statgraph survival_stat;
begingraph;
discreteattrmap name="group" / discretelegendentrypolicy=attrmap;
value "ALL" /lineattrs=GraphData1(pattern=solid thickness=2);
value "AML-Low Risk" / lineattrs=GraphData2(pattern=solid thickness=2);
value "AML-High Risk" / lineattrs=GraphData3(pattern=solid thickness=2);
enddiscreteattrmap;
discreteattrvar var=stratum attrmap="group" attrvar=_grp;
layout overlay /
xaxisopts=(label="Time"
linearopts=(viewmin=0 viewmax=2750
tickvaluesequence=(start=0 end=2750 increment=250)
)
)
yaxisopts=(label="survival probability"
linearopts=(viewmin=0 viewmax=1
tickvaluesequence=(start=0 end=1 increment=0.25)
)
);
/*生存曲線*/
stepplot x=time y=survival /
name="surv"
group=_grp;
/*打ち切り*/
vectorplot x=time y=eval(ifn(censored^=., censored+0.02, .))
xorigin=time yorigin=censored /
group=_grp
arrowheads=false;
/*生存時間中央値テーブル*/
layout overlay / width=40pct height=20pct halign=right
valign=top pad=10px walldisplay=(outline)
opaque=false
yaxisopts=(display=none reverse=true)
xaxisopts=(display=(ticks tickvalues));
innermargin/align=left;
axistable y=NO value=group /
valueattrs=(size=10)
labelattrs=(size=10 weight=bold)
pad=(right=40);
axistable y=NO
value=stat
valueattrs=(size=10)
labelattrs=(size=10 weight=bold);
endinnermargin;
endlayout;
entry "| : Censored " /
autoalign=(topright bottomright)
textattrs=(size=10)
border=true pad=5;
discretelegend "surv" /
location=outside ;
endlayout;
endgraph;
end;
run;
ods graphics /reset;
proc sgrender data=plotdata2 template=survival_stat;
label stat="50% survival (95% CI)";
run;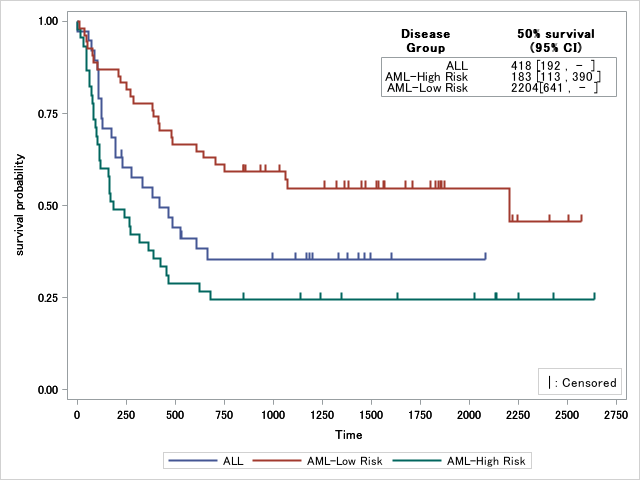
ログランク検定結果を表示する
odsでHomTestsを参照するとログランク検定などの検定結果をデータセットとして取得することができるので、これを作図データセットにマージし、上記の同様に
検定結果を作図することができます。ログランク検定の結果のみを載せたい場合はログランク検定結果のみを抽出してマージすればよいでしょう。
この方法であれば任意の統計量テーブルを好きな場所に配置できるので便利ですね。
proc format;
invalue $bmtifmt 'ALL' = 1
'AML-Low Risk' = 2
'AML-High Risk' = 3;
value bmtfmt 1 = 'ALL'
2 = 'AML-Low Risk'
3 = 'AML-High Risk';
run;
data Bmt2;
set sashelp.BMT(rename=(Group=G));
Group = input(input(G, $bmtifmt.), 1.);
label Group = 'Disease Group';
format Group bmtfmt.;
run;
ods output survivalplot=plotdata
HomTests=test;
proc LIFETEST data=Bmt2 plots=survival(atrisk(maxlen=24)=0 to 2750 by 250 outside) ;
time T*Status(0);
strata Group ;
run;
*データをマージする;
data plotdata3;
merge plotdata test;
run;
proc template;
define statgraph survival2;
begingraph;
discreteattrmap name="group" / discretelegendentrypolicy=attrmap;
value "ALL" /lineattrs=GraphData1(pattern=solid thickness=2);
value "AML-Low Risk" / lineattrs=GraphData2(pattern=solid thickness=2);
value "AML-High Risk" / lineattrs=GraphData3(pattern=solid thickness=2);
enddiscreteattrmap; discreteattrvar var=stratum attrmap="group" attrvar=_grp;
layout overlay /
xaxisopts=(label="Time" type=linear
linearopts=(viewmin=0 viewmax=2750
tickvaluesequence=(start=0 end=2750 increment=250)
)
)
yaxisopts=(label="survival probability"
type=linear
linearopts=(viewmin=0 viewmax=1
tickvaluesequence=(start=0 end=1 increment=0.25)
)
);
/*生存曲線*/
stepplot x=time y=survival /
name="surv"
group=_grp;
/*打ち切り*/
vectorplot x=time y=eval(ifn(censored^=., censored+0.02, .))
xorigin=time yorigin=censored /
group=_grp
arrowheads=false;
/*検定結果*/
layout overlay / width=30pct height=20pct
halign=right valign=top
pad=10px walldisplay=(outline)
opaque=false
yaxisopts=(display=none reverse=true)
xaxisopts=(display=(ticks tickvalues));
innermargin / align=left;
axistable y=test value=Test /
valueattrs=(size=10)
pad=(right=40px)
labelattrs=(size=10 weight=bold);
axistable y=test value=Probchisq/
valueattrs=(size=10)
valuejustify=left
labelattrs=(size=10 weight=bold);
endinnermargin;
endlayout;
/*凡例*/
entry "| : Censored " /
autoalign=(topright bottomright)
textattrs=(size=10)
border=true pad=5;
discretelegend "surv" /
location=outside ;
endlayout;
endgraph;
end;
run;
proc sgrender data=plotdata3 template=survival2;
label Probchisq="P-value";
run;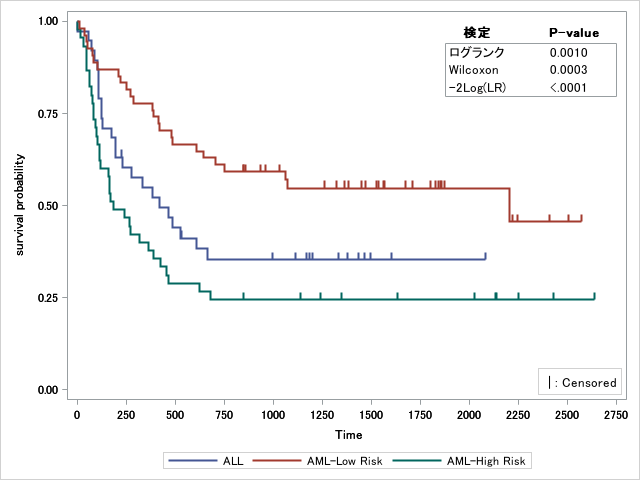
参照線を挿入する
生存率が50%となる時間を参照線を引いてわかりやすくします。
y=0.5の平行線を引き、各層の50%生存率となった時点からX軸にむけて垂線を引きます。
参照線はreferencelineステートメントおよびdroplineステートメントで作図することができます。


/*50%生存率に達するのに要した時間*/
dropline x=estimate y=pct / dropto=x lineattrs=(color=black pattern=shortdash thickness=1);
/*50%生存率*/
referenceline y=0.5 / lineattrs=(color=black thickness=1);「生存時間中央値を表示する」で使用したテンプレートのlayout overlayブロック内に参照線のコードを追加し、再実行すると以下のようになります。