私の昔の勉強していた時のメモが出てきたのでブログネタにします。
生存関数の推定値と信頼区間
ods outputの”Productlimitestimates”から取得できます。任意の時点の推定値が欲しい場合は
timelistオプションで時点を指定します。指定すると変数「Timelist」が追加されます。「Survival」に指定した時点における生存率の推定値が格納されます。
ods output productlimitestimates = estimate ;
proc lifetest data=sashelp.bmt outsurv=test timelist=(0 to 2000 by 500);
time T*status(0);
strata group;
run;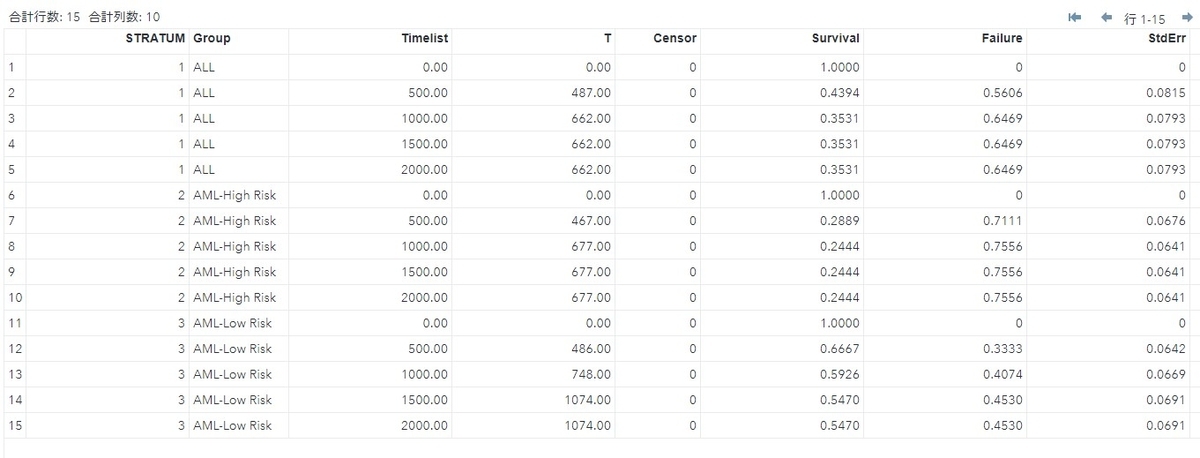
信頼区間も欲しい場合はoutsurvオプションを利用すると取得できます。「SDF_UCL」は信頼上限、「SDF_LCL」は信頼下限です。
proc lifetest data=sashelp.bmt outsurv=cl ;
time T*status(0);
strata group;
run;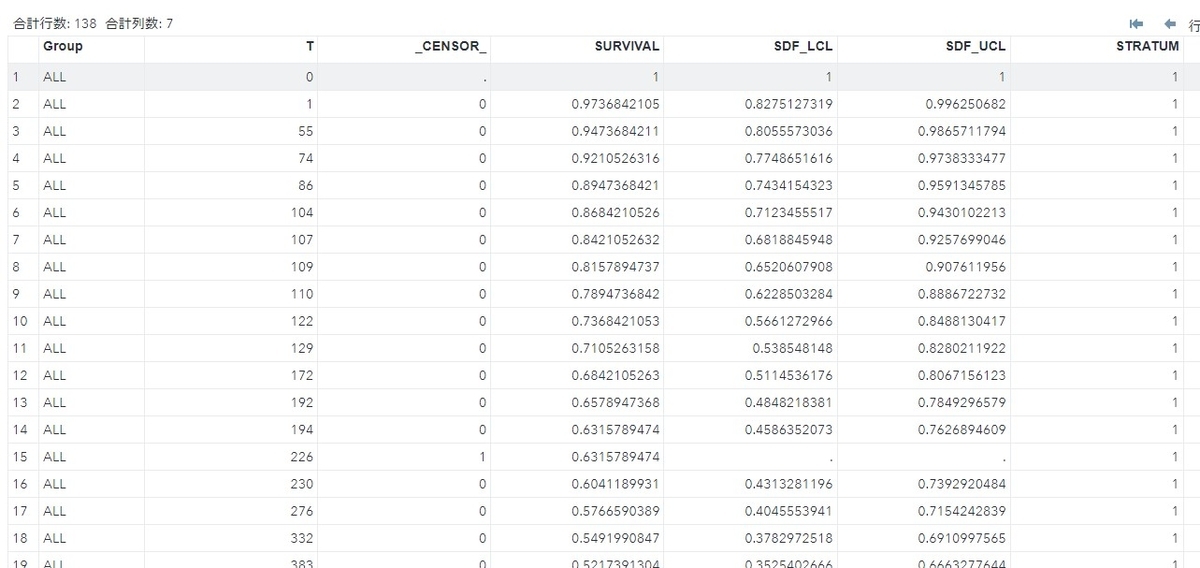
打ち切りの要約
各層の総打ち切り数、総イベント数はods outputの”Censored summary”から取得できます。
ods output censoredsummary=censor;
proc lifetest data=sashelp.bmt;
time T*status(0);
strata group;
run;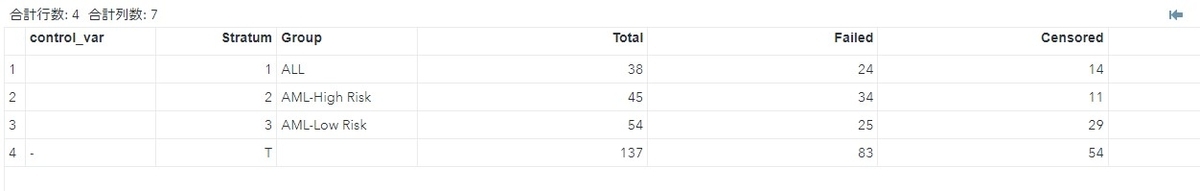
Number at risk
各時点のリスク集合の大きさ(number at risk)を求めます。
Number at risk = 被験者数 - その時点の直前までに発生したイベント数 – その時点の直前までに発生した打ち切り数
number at riskを出力するには proc lifetest にatriskキーワードを追加する必要があります。
出力結果はods outputの”ProductLimitEstimates”の変数「NumberAtRisk」から取得できます。
ods output productlimitestimates =stat1;
proc lifetest data=sashelp.bmt atrisk;
time T * status(0);
strata group;
run;同じデータセットの変数「Left」をNumber at riskとして取得している人がたまにいますが、これはNumber at riskではありません。
生存時間解析でnumber at riskを表示しない事例はまずないので、atriskキーワードは必ず入れておくと憶えておいてよいでしょう。
任意の時点のnumber at riskを取得したい場合はtimelistオプションを使用します。
等間隔の時点の場合は「開始時点 to 終了時点 by 間隔」の構文で指定できます。
時点が等間隔ではない場合はリスト形式「(時点1 時点2 時点3 ……..)」で指定できます。
ods output productlimitestimates =stat1;
proc lifetest data=sashelp.bmt atrisk timelist=(0 to 2000 by 250);
time T * status(0);
strata group;
run;パーセンタイル
生存時間のパーセンタイルおよびその信頼区間はods outputの”quartiles”から取得できます。
推定値は変数「Estimate」、信頼下限および信頼上限はそれぞれ「LowerLimit」および「UpperLimit」から取得しましょう。
信頼区間の有意水準はalphaqtオプションで指定できます。
ods output quartiles = q;
proc lifetest data=sashelp.bmt atrisk ;
time T * status(0);
strata group;
run;デフォルトでは75, 50, 25%点の点推定値を求めることができますが、それ以外のパーセンタイルは求めることはできません。
なので手動で求めるしかありません。以下の論文では任意のパーセンタイルの推定値を求めるマクロが紹介されています。
このマクロだと時間変数や打ち切り変数が固定なので少し修正しました。LOGLOG以外の変換方法もサポートされていますが、
基本的にLOGLOGかLOGITくらいしか使わないのでデフォルトでLOGLOGにしています。
*任意のパーセンタイルを推定する;
* indata=入力データ
persctr=推定したいパーセンタイル
alphapt=有意水準
conftype=変換方法(デフォルトはLOGLOG)
strata = 層変数
time = 時間変数
censor = 打ち切り変数(打ち切りを示す値) ;
%macro ptile(indata=, percstr=, alphapt=0.05, conftype=LOGLOG, strata=, time=, censor=);
** process the percstr ** ;
data perc; length percentile $3.;
perc="&percstr";
num = countw(perc,',');
if num = 0 then do;
percentile = perc; output;
end;
else do k = 1 to num;
percentile = scan(perc,k,','); output;
end;
run;
data _null_;
set perc end=eof;
i+1;
call symput(compress('percentile'||put(i,best.)), trim(left(percentile)));
if eof then call symput('total', put(_n_, best.));
run;
**preparing of Input data;
%if &strata eq %then %do;
%let strata = addstra;
data input_life;
set &indata;
addstra = 1;
run;
%end;
%else %do;
data input_life;
set &indata;
run;
%end;
proc sort data = input_life; by &strata; run;
*survival analysis;
ods output ProductLimitEstimates=km_curv quartiles=quartiles;
proc lifetest data=input_life conftype=&conftype alphaqt=&alphapt method=KM;
strata &strata;
time &time.* &censor.;
run;
** consider five different transformations ** ;
%do k = 1 %to &total;
%if %sysfunc(upcase(&conftype)) = LOGLOG %then %do;
data km;
set km_curv;
if survival not in (.,1) then do;
a1 = log((-1)*log(survival)) - log((-1)*log(%sysevalf(1 - &&percentile&k / 100)));
a2 = stderr / (survival*log(survival));
tranform=a1/a2;
end;
run;
%end;
%else %if %sysfunc(upcase(&conftype))=LOG %then %do;
data km;
set km_curv;
a1 = log(survival)-log(%sysevalf(1-&&percentile&k/100));
a2 = stderr/survival;
tranform = a1/a2;
run;
%end;
%else %if %sysfunc(upcase(&conftype))=LINEAR %then %do;
data km;
set km_curv;
a1 = survival-%sysevalf(1-&&percentile&k/100);
a2 = stderr;
tranform = a1/a2;
run;
%end;
%else %if %sysfunc(upcase(&conftype))=LOGIT %then %do;
data km;
set km_curv;
a1 = log(survival/(1-survival))-
log(%sysevalf(1- &&percentile&k/100)/%sysevalf(&&percentile&k/100));
a2 = stderr/(survival*(1-survival));
tranform = a1/a2;
run;
%end;
%else %if %sysfunc(upcase(&conftype))=ASINSQRT %then %do;
data km;
set km_curv;
a1 = arsin(sqrt(survival)) -
arsin(sqrt(%sysevalf(1 - &&percentile&k / 100)));
a2 = stderr / (2*sqrt(survival)*sqrt(1 - survival));
tranform = a1/a2;
run;
%end;
data km;
set km;
where censor=0;
* 0 is for event;
if (-1)*probit(%sysevalf(1-&alphapt/2)) <= tranform <= probit(%sysevalf(1-&alphapt/2)) then
flag = 'Y';
obs=_n_;
run;
** get the lower limit ** ;
data km1(keep=&strata lower);
set km;
where flag =' Y';
by &strata;
if first.&strata;
lower = &time.;
** get the upper limit **;
data km2(keep=&strata obs);
set km;
where flag = 'Y';
by &strata;
if last.&strata;
obs = obs+1;
data km3(keep = &strata upper);
merge km km2(in = a);
by &strata obs;
if a; upper = &time.;
run;
** get the point estimate ** ;
data calcptiles(keep=&strata ptiles);
set km_curv(where=(censor eq 0));
* 0 is for event;
by &strata;
cutoff = %sysevalf(&&percentile&k/100);
retain _survival ptiles exact;
if first.&strata then do;
ptiles = .;
exact = .;
_survival = .;
end;
if survival > .z then _survival = survival;
if .z < round(_survival,10**-12) <= 1-cutoff then do;
ptiles = min(&time., ptiles);
if exact = 1 then do;
exact = .;
ptiles = (&time.+ptiles)/2;
end;
if round(survival,10**-12) = 1-cutoff then
exact=1;
end;
if last.&strata;
run;
data final&&percentile&k;
merge km1 km3 calcptiles;
by &strata;
percent = "&&percentile&k";
transform = "&conftype";
run;
%if &k = 1 %then %do;
data final;
set final&percentile1;
run;
%end;
%else %do;
data final;
set final final&&percentile&k;
run;
%end;
%end;
proc report data=final; columns &strata percent ptiles transform lower upper;
define &strata/"Stratum";
define percent/"Percent";
define ptiles/"Estimate";
define transform/"Transform";
define lower/"LowerLimit";
define upper/"UpperLimit";
run;
%mend ptile;sashelpのテストデータを使って10, 20, 40, 60, 90 %点を推定してみます。
%ptile(indata=sashelp.bmt,
percstr=%str(10, 20, 40, 60, 90),
alphapt=0.05,
conftype=LOGLOG,
strata=group,
time=T,
censor=status(0));結果は以下の通りです。
| Stratum | Percent | Estimate | Transform | LowerLimit | UpperLimit |
|---|---|---|---|---|---|
| ALL | 10 | 86 | LOGLOG | 1 | 110 |
| AML-High Risk | 10 | 47 | LOGLOG | 2 | 74 |
| AML-Low Risk | 10 | 80 | LOGLOG | 10 | 248 |
| ALL | 20 | 110 | LOGLOG | 74 | 192 |
| AML-High Risk | 20 | 75 | LOGLOG | 47 | 105 |
| AML-Low Risk | 20 | 272 | LOGLOG | 79 | 481 |
| ALL | 40 | 276 | LOGLOG | 122 | 487 |
| AML-High Risk | 40 | 138.5 | LOGLOG | 84 | 268 |
| AML-Low Risk | 40 | 748 | LOGLOG | 414 | . |
| ALL | 60 | 609 | LOGLOG | 332 | . |
| AML-High Risk | 60 | 340.5 | LOGLOG | 162 | 677 |
| AML-Low Risk | 60 | . | LOGLOG | 2204 | . |
| ALL | 90 | . | LOGLOG | . | . |
| AML-High Risk | 90 | . | LOGLOG | . | . |
| AML-Low Risk | 90 | . | LOGLOG | . | . |
念のためRを使って検算をします。
Rには生存時間解析パッケージSurvivalがありますので、これのsurvfit関数を使用します。データセットはあらかじめexcelファイルにエクスポートしています。
library(tidyverse)
library(survival)
library(readxl)
a <-read_excel('BMT.xlsx') # カプランマイヤー推定の実施
fit <- survfit(Surv(T, Status) ~ Group, data = a, conf.type="log-log")
# パーセンタイル
quantile(fit, 0.1)
quantile(fit, 0.2)
quantile(fit, 0.4)
quantile(fit, 0.6)
quantile(fit, 0.9)結果は以下の通りです。あってますね。
> quantile(fit, 0.1) $quantile 10 Group=ALL 86 Group=AML-High Risk 47 Group=AML-Low Risk 80 $lower 10 Group=ALL 1 Group=AML-High Risk 2 Group=AML-Low Risk 10 $upper 10 Group=ALL 110 Group=AML-High Risk 74 Group=AML-Low Risk 248 > quantile(fit, 0.2) $quantile 20 Group=ALL 110 Group=AML-High Risk 75 Group=AML-Low Risk 272 $lower 20 Group=ALL 74 Group=AML-High Risk 47 Group=AML-Low Risk 79 $upper 20 Group=ALL 192 Group=AML-High Risk 105 Group=AML-Low Risk 481 > quantile(fit, 0.4) $quantile 40 Group=ALL 276.0 Group=AML-High Risk 138.5 Group=AML-Low Risk 748.0 $lower 40 Group=ALL 122 Group=AML-High Risk 84 Group=AML-Low Risk 414 $upper 40 Group=ALL 487 Group=AML-High Risk 268 Group=AML-Low Risk NA > quantile(fit, 0.6) $quantile 60 Group=ALL 609.0 Group=AML-High Risk 340.5 Group=AML-Low Risk NA $lower 60 Group=ALL 332 Group=AML-High Risk 162 Group=AML-Low Risk 2204 $upper 60 Group=ALL NA Group=AML-High Risk 677 Group=AML-Low Risk NA > quantile(fit, 0.9) $quantile 90 Group=ALL NA Group=AML-High Risk NA Group=AML-Low Risk NA $lower 90 Group=ALL NA Group=AML-High Risk NA Group=AML-Low Risk NA $upper 90 Group=ALL NA Group=AML-High Risk NA Group=AML-Low Risk NA
同等性の検定結果
ログランク検定、一般化wilcoxon検定などの結果はods outputの”homtest”に格納されています。
ods output homtests=test;
proc lifetest data=sashelp.bmt atrisk timelist=(0 to 2000 by 250);
time T * status(0);
strata group;
run;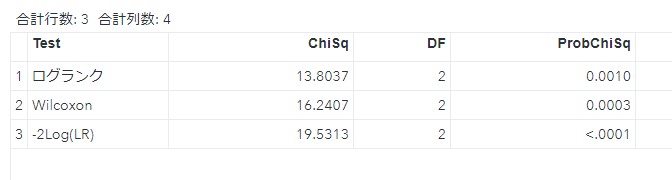
生存曲線データ
生存曲線はplotsオプションでsurvivalを指定し、ods outputの”Survivalplot”から曲線のデータが取得できます。
作図したい場合は自身で前処理するのではなくこのデータを使ったほうが間違いなしです。
詳細は以下の記事で紹介しています。

関連記事もあわせてご確認ください。








