臨床統計においてよく作図されるカプランマイヤープロットの打ち切りの記号は上ひげ(縦棒)を用いられることがよくあります。
しかしSASのLifetestプロシジャのデフォルトではプラス記号を用いており、変更しようにも標準では縦棒のマーカーが用意されておりません。
今回は上ひげを作図する方法をご紹介します。
作図方法
作図方法には以下の4つがあります。
- scatterplotステートメントのmarkercharacterオプションを利用する方法
- symbolcharステートメントで縦棒のカスタムマーカーを作成する方法
- データを加工してseriesplotでひげを作成する方法
- vectorplotステートメントを利用する方法
昔はデータを加工してexcelグラフかseriesplotで作図する方法と、annotation datasetでひげを作成する方法がありましたが、
いずれもひげを作成するためにレコードを生成する必要がありデータの可読性が悪くなります。
anotation datasetは言うまでもなくめんどくさいです。
今回は最も汎用性の高い4の方法で実施します。
vectorplotステートメント
vectorplotステートメントは任意の2点を結ぶ線分を作図するステートメントです。
vectorplot x=< 終点のX座標 > y=< 終点のY座標 >
xorigin= < 始点のX座標 > yorigin= < 始点のY座標 > /< options > ;
このステートメントで打ち切りが発生した時点の生存率の座標から垂直方向へ線分を伸ばすことで上ひげを作成することができます。
この方法の利点は生存率データセットを前処理する必要がない点と、SAS9.3でも動作する点です。
作図コード
それでは実際にSASのサンプルファイルを用いて作図してみます。
作図データセットの変数censoredは打ち切り記号のY座標を格納する変数です。これを利用して線分を作図します。
デフォルトでは矢印を作図するのですが、arrowheads=falseを指定すると矢印の頭が非表示となり、通常の線分として表示されます。
線分の始点は打ち切り記号の位置(X=time, Y=censored)で、終点はそこから垂直に0.02移動した点を指定しています。
eval関数とifn関数を併用すれば、わざわざデータセットに終点のY座標の変数を追加する必要はありません。
ifn関数でcensoredが欠損値でない場合、censoredに0.02を加えた数値を格納した変数をGTL内部で新規作成します。
0.02は上ひげの線分の長さということですね。この数値を変更することでひげの長さを変更することも可能です。
これによりlifetestプロシジャが出力したデータセットをそのまま読み込ませるだけで上ひげの記号を用いた作図が可能となります。
凡例を追加したい場合は、entryステートメントで打ち切り記号の説明を自作する必要があります。
この方法もSAS9.3で動作します。
proc format;
invalue $bmtifmt 'ALL' = 1
'AML-Low Risk' = 2
'AML-High Risk' = 3;
value bmtfmt 1 = 'ALL'
2 = 'AML-Low Risk'
3 = 'AML-High Risk';
run;
data Bmt2;
set sashelp.BMT(rename=(Group=G));
Group = input(input(G, $bmtifmt.), 1.);
label Group = 'Disease Group';
format Group bmtfmt.;
run;
ods output survivalplot=plotdata;
proc LIFETEST data=Bmt2 plots=survival;
time T*Status(0);
strata Group ;
run;
proc template;
define statgraph surv;
begingraph; discreteattrmap name="group" / discretelegendentrypolicy=attrmap;
value "ALL" / lineattrs=GraphData1(pattern=solid);
value "AML-Low Risk" / lineattrs=GraphData2(pattern=solid);
value "AML-High Risk" / lineattrs=GraphData3(pattern=solid);
enddiscreteattrmap;
discreteattrvar var=stratum attrmap="group" attrvar=_grp;
layout overlay /
xaxisopts=(label="Time"
linearopts=(viewmin=0 viewmax=2750
tickvaluesequence=(start=0 end=2750 increment=250)
)
)
yaxisopts=(label="survival probability"
linearopts=(viewmin=0 viewmax=1
tickvaluesequence=(start=0 end=1 increment=0.25)
)
);
/*生存曲線*/
stepplot x=time y=survival /
name="surv"
group=_grp;
/*打ち切り*/
vectorplot x=time y=eval(ifn(censored^=., censored+0.02, .))
xorigin=time yorigin=censored /
group=_grp
arrowheads=false;
/*凡例*/ entry "| : Censored " /
autoalign=(topright bottomright)
textattrs=(size=10)
border=true
pad=5;
discretelegend "surv" /
location=outside ;
endlayout;
endgraph;
end;
run;
proc sgrender data=plotdata template=surv;
run;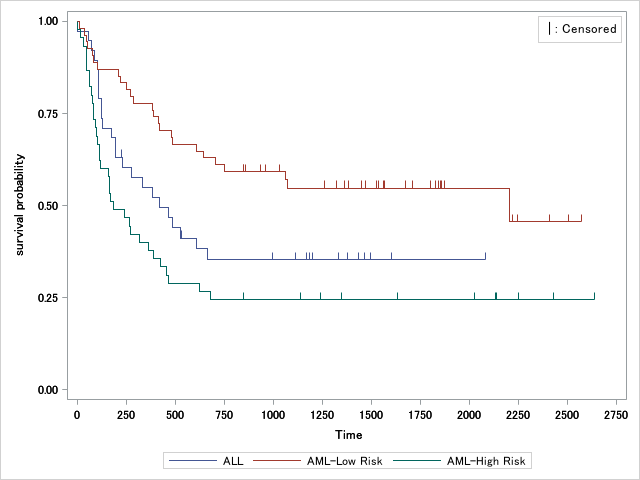
これはかなり有用だと思うのでぜひ使ってみてください。



