「SAS Plotter」はモダンなグラフを簡単に作成できるグラフマクロパッケージです。
このページではSAS Plotter version 1.3でPaied Raincloud plotを作成する方法を解説します。
version 1.3からインストール方法が変更になりました。詳細は以下のページをご参照ください。

- Paired Raincloud plot とは
- Input data
- 文法
- パラメータ
- data : データセット (必須)
- x : 変数 (必須)
- y : 変数(必須)
- repeat : 変数(必須)
- subject : 変数(必須)
- group : 変数(任意)
- ylabel : テキスト(任意)
- yticks : リスト (必須)
- cat_iv : 数値(任意)
- element_iv : 数値(任意)
- scale : キーワード (任意)
- trim : 真偽値 (任意)
- connect : 真偽値(任意)
- spaghetti : 真偽値 (任意)
- gridsize : 数値(任意)
- bw_method : キーワード(任意)
- bw_adjust : 数値 (任意)
- legend : 真偽値(任意)
- legendtitle : テキスト(任意)
- orient : キーワード(任意)
- jitterwidth : 数値(任意)
- outlinewidth : 数値(任意)
- pallete : キーワード (オプション)
- note : ステートメント(オプション)
- deletedata : 真偽値(オプション)
- 作図例
- 専用フォーラム
Paired Raincloud plot とは
Paired Raincloud plotは反復測定データをRainCloud plotで可視化するときに使います。
通常のRaincloudと異なり、個別データの推移をSpgaghetti plotで表現します。またペアとなるデータの密度プロットは左右に反転させて表示します。
反復は2回まで対応している点に注意してください。それ以上の反復データがあると
正常に作図されません。
Input data
| key | 変数 | 変数タイプ |
|---|---|---|
| 1 | カテゴリ変数 | 数値または文字列 |
| 2 | 反復識別変数 | 数値または文字列 |
| 3 | 群変数 | 数値または文字列 |
| 被験者識別変数 | 数値または文字列 | |
| 応答変数 | 数値 |
群変数は任意です。群変数とカテゴリ変数は文字列型でも動作しますが、その場合はテキストの昇順でグループの表示順を決定します。
順番を定義したい場合はフォーマットを適用した数値変数を使用することを推奨します。
文法
ods graphics / < graphics option > ;
ods listing gpath=< output path >;
%RainCloud(
data=,
x=,
y=,
group=None,
yticks=,
xlabel=x,
ylabel=y,
cat_iv=2.5,
element_iv=0.02,
scale=area,
trim=True,
connect=false,
gridsize=401,
bw_method=sjpi,
bw_adjust=1,
orient=v,
legend=false,
legendtitle=#,
jitterwidth=0.1,
outlinewidth=1,
palette=sns,
note=,
deletedata=True);パラメータ
data : データセット (必須)
入力データ。keep, rename, whereオプションを併用できます。
x : 変数 (必須)
カテゴリ変数
y : 変数(必須)
応答変数
repeat : 変数(必須)
反復識別変数、反復は各被験者あたり2回までです。
subject : 変数(必須)
被験者識別変数
group : 変数(任意)
群変数。xオプションと同じ変数を指定すると、各グループはそれぞれ別の色で塗り分けられます。
指定しない場合はすべてのオブジェクトは同じ色になります。
デフォルト=None
ylabel : テキスト(任意)
応答変数軸のラベル。デフォルトはy変数のラベル。以下のように設定するとラベルは非表示となります。
ylabel=,
yticks : リスト (必須)
応答変数軸の目盛りを指定できます。半角スペースで区切られた数値リストを指定してください。数値は昇順で指定する必要があります。
(例)yticks = 10 20 30 40,
cat_iv : 数値(任意)
各カテゴリの間隔。デフォルトは1.6。
element_iv : 数値(任意)
密度プロット、箱ひげ図、ストリッププロット(スパゲッティプロットのマーカー)の間隔。デフォルトは0.4。
scale : キーワード (任意)
密度プロットの高さの調整方法を指定します。
“area”にすると密度プロットの曲線下面積はすべて同じになるように調整されます。
“width”にすると各密度プロットの高さは同じになります。
デフォルトは”area”。
trim : 真偽値 (任意)
Trueに設定すると観測データの範囲外の密度は非表示となります。
デフォルトはTrue。
connect : 真偽値(任意)
Trueに設定すると箱ひげ図の平均値をグループごとに線でつなぎます。
デフォルトはFalse。
spaghetti : 真偽値 (任意)
Trueに設定するとスパゲッティプロットを表示します。各被験者のデータは実線で結ばれます。
デフォルトはTrue
gridsize : 数値(任意)
KDEのグリッド数。デフォルトは401(proc kdeのデフォルト値)
bw_method : キーワード(任意)
KDEのバンド幅推定方法を指定します。デフォルトはsjpi(proc kdeのデフォルト値)。
利用可能な推定方法は以下の通りです。
- sjpi (Sheather-Jones plug-in)
- snr (simple normal reference)
- snrq (simple normal reference that uses the interquartile
range) - srot (Silverman\’s rule of thumb)
- os (oversmoothed)
bw_adjust : 数値 (任意)
バンド幅のファクター。この値を大きくすると密度はより滑らかになります。デフォルトは1。
legend : 真偽値(任意)
Trueを指定すると凡例を表示します。groupオプションを”None”を指定するとこの引数は無視されます。 デフォルトはFalse。
legendtitle : テキスト(任意)
凡例タイトルを指定します。legendパラメータがFalseの場合は無視します。デフォルトはgroup変数のラベル。
orient : キーワード(任意)
作図の方向を指定します。v=垂直方向 h=水平方向。
デフォルトはv。平均値の推移に注目している場合はv、分布形状の比較に注目している場合はhを選択するとよいと思います。
jitterwidth : 数値(任意)
ストリッププロットの揺らぎ幅を指定します。0~1の範囲の数値を指定し、値が大きくなると揺らぎが大きくなります。
値が大きすぎると箱ひげ図と重なるかもしれません。デフォルトは0.05。
outlinewidth : 数値(任意)
密度プロットと箱ひげ図の枠線の太さを数値で指定します。
デフォルトは1。
pallete : キーワード (オプション)
塗り、ライン、マーカーのカラーパレット、以下のパレットが利用可能です。デフォルトはSNS(seabornのデフォルトパレット)
- SAS
- SNS (Seaborn)
- STATA
- TABLEAU
note : ステートメント(オプション)
グラフテンプレートにentryステートメントを挿入し、グラフタイトルまたはフットノートを表示します。デフォルトは””(表示しない)
deletedata : 真偽値(オプション)
マクロが作成した一時データを削除します。デフォルトはTrue(一時データを削除する)。
作図例
作図例はgithubのexample内にsasプログラムを格納しています。filenameステートメントを使えばすぐに実行できます。
ods listing gpath=<your output path>;
filename exam url "https://github.com/Superman-jp/SAS_Plotter/raw/main/example/raincloudpaired_example.sas" encoding="UTF-8";
%include exam;シンプルなプロット
本マクロは繰り返しのある2群間比較を実施する試験に適しています。特に2*2クロスオーバー試験には相性抜群です。まず架空のクロスオーバー試験のサンプルデータを作成します。
proc format;
value repeatf
1="period 1"
2="period 2";
value seqf
1="sequence A (placebo to drug A)"
2="sequence B (drug A to lacebo)"
;
value trtf
1="Placebo"
2="Drug A"
;
value groupf
1="Factor XXX (-)"
2="Factor XXX (+)";
run;
data raincloudtest;
call streaminit(1234);
format repno repeatf. trt seqf.;
label response="activity";
do trt=1 to 2;
do i=1 to 100;
do repno=1 to 2;
usubjid="A" || strip(put(i,z3.0));
if trt=1 and repno=1 then response=rand("lognormal",3,0.2);
else if trt=1 and repno=2 then response=rand("lognormal",3.5,0.23);
else if trt=2 and repno=1 then response=rand("lognormal",3.4,0.21);
else if trt=2 and repno=2 then response=rand("lognormal",2.8,0.17);
if response < 0 then response=0;
output;
end;
end;
end;
run;まずはシンプルな繰り返しのあるraincloud plotを作成してましょう。
title " paired raincloud plot (vertical)";
ods graphics / reset=all imagefmt=png imagename="rainpair_simple_v" width=20cm height=20cm;
%RainCloudPaired(
data=raincloudtest,
x=trt,
y=response,
group=trt,
repeat=repno,
subject=usubjid,
orient=v,
yticks= 0 20 40 60 80,
note=%nrstr(entrytitle 'your title here';
entryfootnote halign=left 'your footnote here';
entryfootnote halign=left 'your footnote here 2';)
);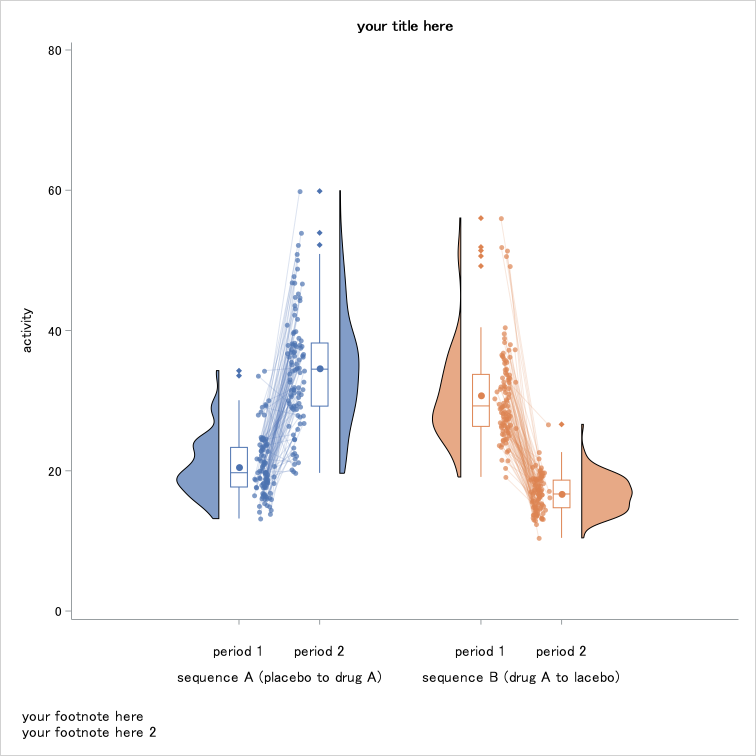 クロスオーバー試験結果の可視化例(垂直方向にプロット)
クロスオーバー試験結果の可視化例(垂直方向にプロット)orientパラメータをhにするとプロットは水平方向に変更されます。
(x軸=応答変数, y軸=カテゴリ)
title "paired raincloud plot (horizontal)";
ods graphics / reset=all imagefmt=png imagename="rainpair_simple_h" width=20cm height=20cm;
%RainCloudPaired(
data=raincloudtest,
x=trt,
y=response,
group=trt,
repeat=repno,
subject=usubjid,
orient=h,
scale=width,
yticks= 0 20 40 60 80,
note=%nrstr(entrytitle 'your title here';
entryfootnote halign=left 'your footnote here';
entryfootnote halign=left 'your footnote here 2';)
);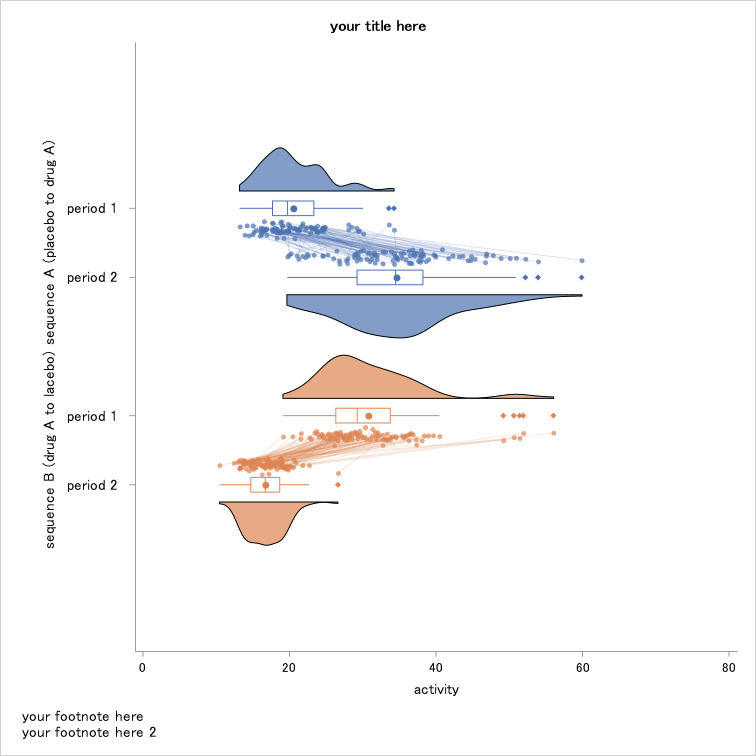 クロスオーバー試験結果の可視化例(水平方向にプロット)
クロスオーバー試験結果の可視化例(水平方向にプロット)グループ毎に表示
次はFactor XXXの有無別にplaceboとDrug Aのlog(AUC)をプロットしてみましょう。
rawデータ
data raincloudtest2;
call streaminit(1234);
format trt trtf. type groupf.;
label response="log(AUC)" cat="population";
cat="FAS";
do type=1 to 2;
do i=1 to 100;
do trt=1 to 2;
usubjid="A" || strip(put(i,z3.0));
if type=1 and trt=1 then response=rand("normal",0.8,0.15);
else if type=1 and trt=2 then response=rand("normal",1.3,0.25);
else if type=2 and trt=1 then response=rand("normal",1.0,0.17);
else if type=2 and trt=2 then response=rand("normal",2.1 ,0.22);
output;
end;
end;
end;
run;);
title "paired raincloud plot (grouped)";
ods graphics / reset=all imagefmt=png imagename="rainpaird_group" width=20cm height=15cm;
%RainCloudPaired(
data=raincloudtest2,
x=cat,
y=response,
group=type,
repeat=trt,
subject=usubjid,
orient=v,
legend=true,
yticks=0 0.5 1 1.5 2 2.5 3.0
);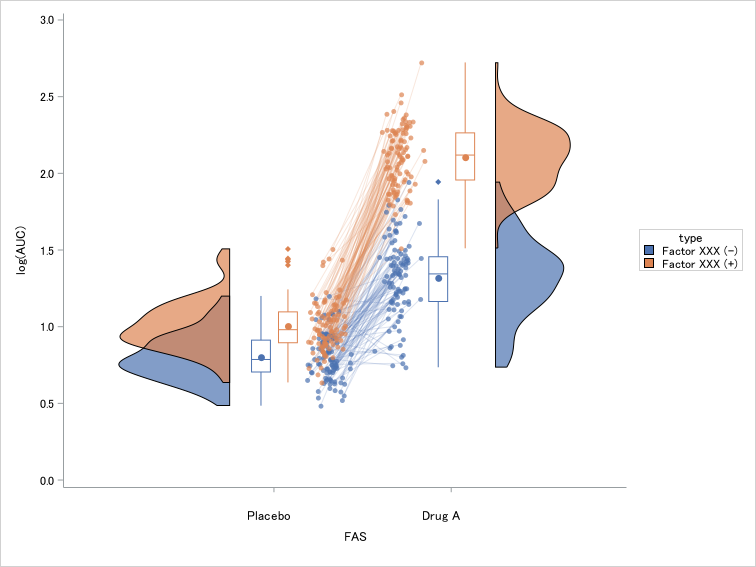 Factor XXXの有無別log(AUC)分布状況
Factor XXXの有無別log(AUC)分布状況Connect パラメータ
connectパラメートをTrueに設定すると、各グループの平均値はラインで結ばれます。
スパゲッティプロットとは独立しているため、スパゲッティプロットの上に作図することも可能です。
title "paired raincloud plot with connect line";
ods graphics / reset=all imagefmt=png imagename="rainpaird_group_connect" width=20cm height=15cm;
%RainCloudPaired(
data=raincloudtest2,
x=cat,
y=response,
group=type,
repeat=trt,
subject=usubjid,
orient=v,
legend=true,
connect=true,
yticks=0 0.5 1 1.5 2 2.5 3.0
);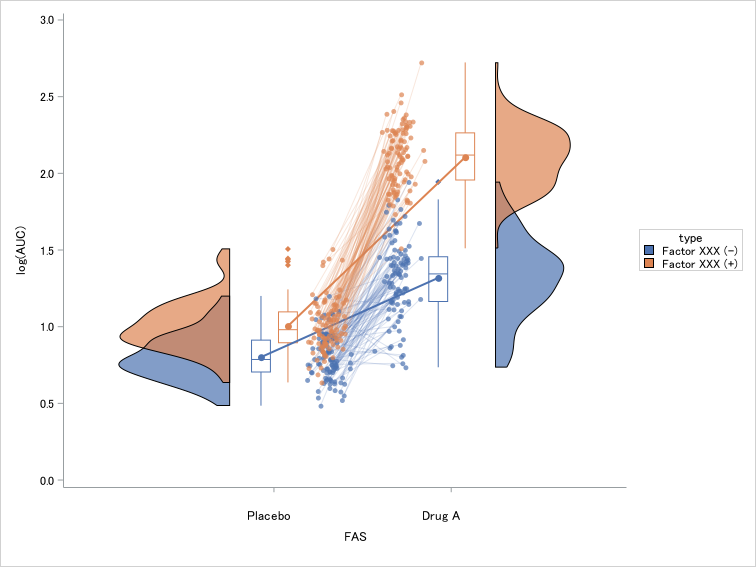 平均値を線でつなぐ
平均値を線でつなぐscaleパラメータ、およびtrimパラメータは通常の%RainCloudと同じ挙動です。
詳細はRaincloud plotの紹介記事を参照してください。
https://picolabs.jp/SASPlotter_Raincloud
専用フォーラム
要望、バグ報告は以下のフォーラムをご利用ください






